频率域中生物进化树的构建及其应用毕业论文
2020-07-08 21:51:11
摘 要
DNA序列相似性研究已经成为人类探索生命之起源,揭开生命奥秘的必经之路,本文主要介绍了DNA序列相似性研究的理论知识和相关应用,并提出了对未来的展望。
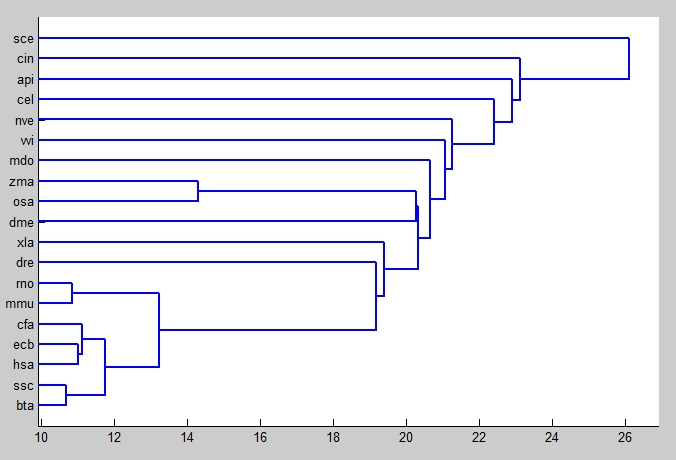
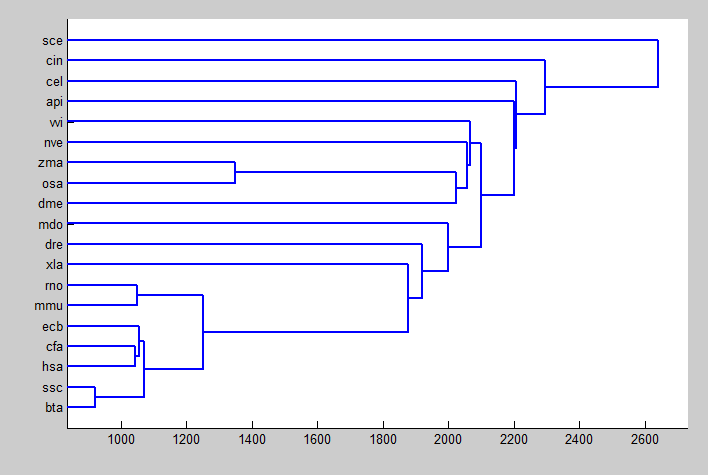
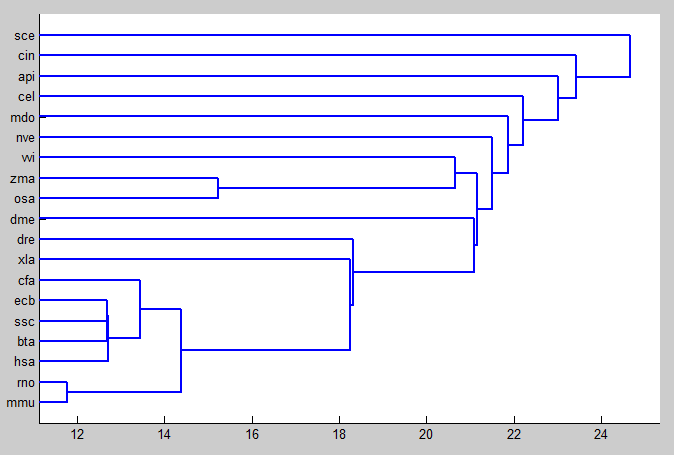
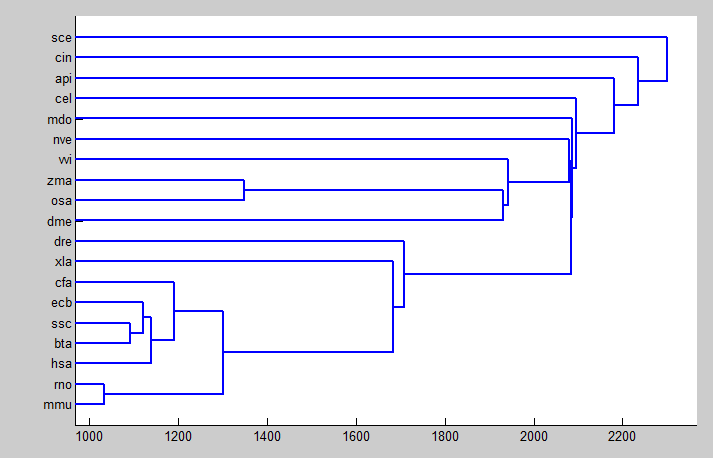
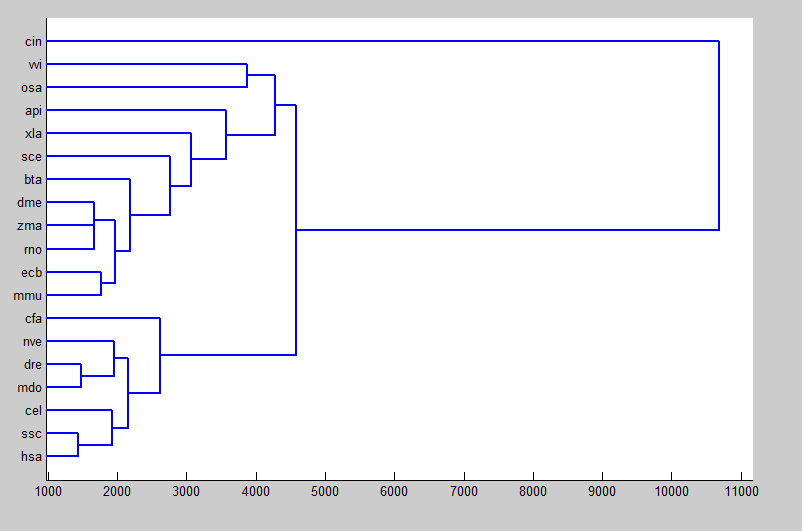
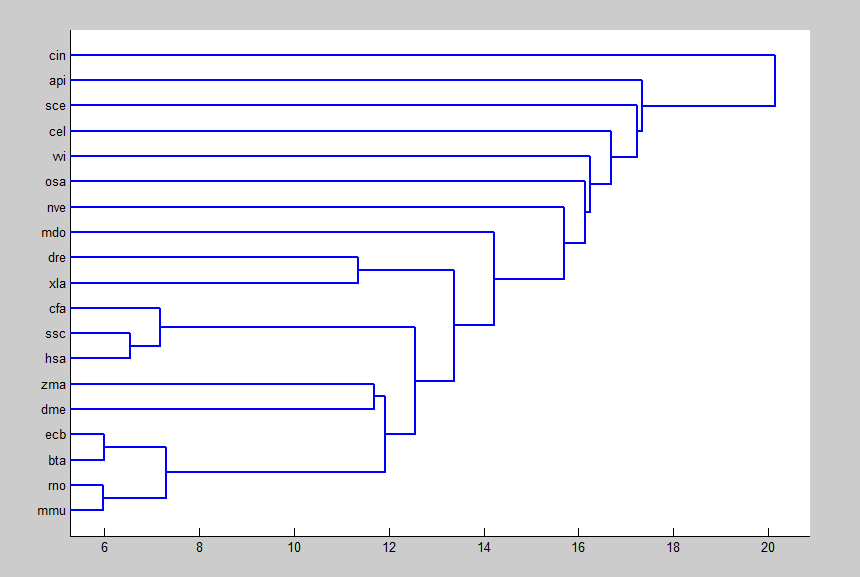
在探索DNA序列相似性过程中首先需要通过信号处理将DNA序列转换成数字表示,针对目前已经被提出的几种数字表示法,本文采用了不同维度的三种映射方法得到不同的数字序列后,通过应用快速傅立叶变换使数字序列得以在频率域上提取等长的特征序列,在此基础上,分别采用了欧氏距离,归一化欧氏距离和曼哈顿距离三种度量进行聚类分析,生成聚类树,分析生物序列之间的相似性。本文从基因数据库中下载对应于核糖体蛋白编码基因RP-S18 的DNA序列,构建序列聚类树图。
文章最后简要评价该方法的优缺点并对今后DNA相似性的研究及应用提出展望。
关键词: DNA相似性 DNA数字表示法 快速傅立叶算法 功率谱 聚类分析
Abstract
The research of DNA sequence similarity has become the only way to study the origin of human and expose the mystery of life. This paper mainly introduces the theoretical knowledge and related applications of DNA sequence similarity research, and finally puts forward the prospect of the future.
In the process of exploring DNA sequence similarity, we first need to convert the DNA sequence into a digital representation through signal processing. For the several digital representations that have been proposed so far, this paper uses three mapping methods of different dimensions to obtain different digital sequences. By applying Fast Fourier Transform, the digital sequence can extract feature sequences of equal length in the frequency domain. On this basis, Euclidean distances, normalized Euclidean distances and Manhattan distances are used for cluster analysis and generating Cluster trees to analyze the similarity between biological sequences. This article downloads the DNA sequence corresponding to the ribosomal protein encoding gene RP-S18 from the gene database and constructs a sequence clustering tree map.
At the end of the article, the advantages and disadvantages of the method are briefly evaluated, and the future of DNA similarity research and application are prospected.
Key Words:DNA similarity; DNA digital representation; Fast Fourier algorithm; Power spectrum; Cluster Analysis
目 录
摘 要 I
Abstract II
第一章 绪论 1
1.1 研究背景和意义 1
1.2 DNA序列背景知识 1
1.2.1 碱基互补配对原则 1
1.2.2编码蛋白质 2
1.3 本文主要工作 2
第二章 DNA序列信号处理方法 3
2.1 DNA序列数值表示法 3
2.1.1 实数映射法 3
2.1.2 VOSS映射法 4
2.1.3 “Z曲线表示”映射法 4
2.2 DNA序列信号分析与处理 5
2.2.1 离散傅立叶变换 5
2.2.2 快速傅立叶变换 6
2.2.3 功率谱密度计算 7
第三章 序列相似性度量标准 8
3.1欧氏距离与归一化欧氏距离 8
3.2曼哈顿距离 9
3.3 序列相似性应用实例 9
3.3.1 聚类树的生成 9
第四章 结语与展望 15
参考文献 16
第一章 绪论
1.1 研究背景和意义
1953年,DNA的双螺旋结构在人们的不懈探索下终于被杰出的科学家所发现,沃森和克里克仅用了一些简单部件搭建的DNA分子模型却成了开启生命研究新领域的钥匙。自这层迷糊了生命科学界几十年的窗户纸被捅破后,人们对生命繁衍的研究如雨后春笋一般获得了一个接一个的突破,生物信息数据库日益丰富起来,极大程度上推进了生命科学的发展进程。与此同时,计算机技术的发展成了科学研究的中流砥柱,计算机也就成了被广泛使用的对生物数据进行储存,探索,整理和分析的关键工具。随着生命科学与计算机科学愈加密切的结合与发展,生物信息学这门新学科也就由此诞生了。而DNA序列比对是生物信息学的基本组成和重要基础,它的理论基础是进化学说。检测DNA序列之间的相似性,是探究物种同源性的关键。如果某两条DNA序列之间的相似性足够高并具有显著的保守性,可推证二者可能有共同的进化祖先,为确保实验的偶然性与严谨性,我们依然还需要进行大量的实验以及挖掘更多的信息,去探索二者是否具有类似的序列结构和功能特点。
随着人们对生物信息的不断挖掘,数据如源源不断的溪流通过互联网从世界四面八方汇聚成一片汪洋,人们通过丰富多样的数据分析处理方法来对生物信息数据库进行整理,研究和分析,这些数据的功能和研究意义一直在不断开发中。其中,序列之间的相似性是几年来一直热门的研究领域。除了可以构建生物进化树探索物种起源,还可以用于克隆疾病基因,生物制药,基因诊断并治疗以及预防遗传病,故而研究意义重大。
生命在进化过程中,既会延续上一代的特征,又会随着环境的影响产生新的特征,故而不能保证百分百的复制。DNA序列可能会经历缺失,替换,重组等遗传变异过程,遗传特征也因此被删除或改变,于是,后代才会有别于亲代,才使得生命得到进化。因此用来比对的DNA序列大多长短不一,针对不等长序列的相似性的研究往往更加具有重要的现实意义和实践意义。
相关图片展示:

课题毕业论文、开题报告、任务书、外文翻译、程序设计、图纸设计等资料可联系客服协助查找。



